PoSeNoGap
Portable Scalable Concurrency for Genomic Data Processing
- Responsable
- THOMA Yann
- Période
-
février 2014 -
juin 2017
-
Opening existing...

- Axes
-


- Domaines
-

PoSeNoGap vise à développer un nouveau framework pour traitement des données génomiques. A l'heure actuelle le séquençage s'un génome humain génère environ 300GB de données brutes. Dès lors il est indispensable de pouvoir les compresser, et l'analyse de ces données est très coûteuse en temps de traitement. Durant les trois ans de PoSeNoGap, de nouvelles méthodes de compression sont en cours de développement. Dans ce contexte, l'institut REDS développe une solution performante pour le clustering de séquences non mappées grâce à une plateforme FPGA de PicoComputing.
Avec l'avènement des nouvelles technologies de séquençage haut-débit, la récupération de l'information du génome d’un individu est devenu possible et abordable. Ces méthodes ouvrent des approches inédites dans le cadre du diagnostique et du traitement efficace du cancer et des maladies génétiques. Il reste cependant de nombreux défis de nature scientifique et informatique à relever avant de pouvoir intégrer ces technologies émergentes à l'infrastructure médicale existante. Un premier défi consiste à gérer le déluge de données généré (Pour ordre de grandeur, plusieurs téraoctets de données sont collectées chaque semaine rien que par le centre Vital-IT du SIB). Une base de données représentative pour les habitants d'un petit pays comme la Suisse, aurait besoin de stocker une quantité immense de données, plus de 2'000'000 de téraoctets. Un second défi est lié à la puissance de calcul nécessaire afin de tirer profit de ces données. Pour 1) Consolider les connaissances scientifiques sur l'information contenue dans la séquence du génome et 2) Parcourir les bases de données dans le but de diagnostiquer et de traiter des patients. Compresser de manière significative ces données est nécessaire afin de rendre possible la conservation entière de celles-ci mais aussi pour réduire la bande passante nécessaire à la transmission des séquences génomiques entre les bases de données les hôpitaux et les centres de recherches. Afin de pouvoir traiter cette quantité astronomique d'informations en un temps raisonnable les algorithmes utilisés doivent employer des solutions novatrices et massivement parallèles. Leur implémentation doit pouvoir s'adapter au matériel existant et futur afin de réduire toujours le temps nécessaire à l'obtention de résultats.
Le but de ce projet est de développer, pour la "Swiss Platform for Advanced Scientific Computing", un nouveau noeud de calcul composé de matériel hétérogène, un nouveau format de compression des données génomiques ainsi qu'une infrastructure logicielle permettant aux applications émergentes, comme par exemple l'analyse du génome (RNASeq, séquençage de l'ARN), de traiter des quantités extrêmement vastes de données génomiques et ceci dans un temps acceptable. Le temps requis pour obtenir des résultats est souvent critique tant dans le domaine de la recherche scientifique que pour le diagnostique de patients (application clinique) comme présenté dans la Figure 1. Une application dans le domaine RNASeq (p.ex. tophat, bowtie ou aaplication similaire) sera choisie afin de démontrer l'impact d'une implémentation matérielle et logicielle haute performance et servira d'exemple pour d'autres applications.
Pour atteindre son but le projet devra proposer une solution efficace aux problèmes suivants :
- Tirer parti de manière intégrante des application existantes telles que tophat et bowtie pour l'analyse du génome
- Compression et décompression des données
- Trouver parmi les algorithmes de compression existant ceux, qui non seulement permettent un bon taux de compression mais permettent une mise en oeuvre efficace des algorithmes d’analyse sur ces données compressées.
- Sur la base des résultats de compression et d'efficacité des algorithmes existants élaborer de nouveaux algorithmes plus performants.
Moteurs de calcul :
- Le développement des différentes parties logicielles nécessaires à l'analyse et l'accès des données génomiques compressées sera réalisé à l'aide de méthodologies adaptables de programmation en flux de données haut niveau.
- Un ensemble d'outils permettant la synthèse et le profilage de programmes flux de données haut niveau sera développé afin de pouvoir créer des applications exploitant de la manière la plus efficace possible l'infrastructure de traitement et de calcul du PASC.
- Des librairies spécifiques seront développées dans le but de supporter des implémentations d’algorithmes supplémentaires.
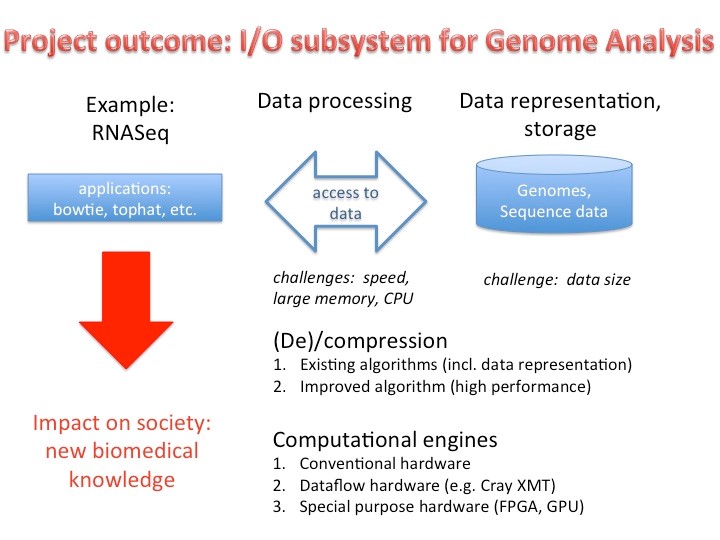
Ce projet vise à relever les défis suivants :
- Rechercher et réaliser des moyens de transfert très haut débit du génome tout en incorporant directement l'algorithme de compression et décompression à l'aide de coprocesseurs matériels. La nature séquentielle des données génomiques et sa représentation sous forme de nucléotides (bases) rend l'utilisation de processeurs conventionnels inefficace et nécessite donc une architecture spécialisée et optimisée afin d'être compressée de manière rapide et efficace. Une architecture sera proposée et débattue avec l'équipe du CSCS (Centre Suisse du Calcul Scientifique) afin de pouvoir intégrer leur future plateforme de calcul
- Refactoriser les algorithmes d'analyse et de traitement génomique actuels et leur applications afin de pouvoir tirer parti du paradigme de programmation en flux de données haut niveau et du parallélisme résultant. Ce travail est fondamental non seulement pour atteindre des niveaux de performance élevés mais aussi afin de pouvoir exploiter un matériel toujours en évolution, basé sur des technologies toujours plus récentes et bénéficiant toujours de nouvelles capacités. Jusqu'à présent l'apparition de nouvelles technologies demandait d'adapter et de porter les algorithmes existants et ceci pour chaque nouvelle architecture afin de tirer pleinement parti des performances offertes. La portabilité d'un langage de programmation en flux de données haut niveau permet d'éviter de devoir changer les algorithmes et applications existantes lors de l'apparition nouvelles technologies avec de nouveaux ou plus grand niveaux de parallélisme. Refactoriser le code existant permettra de créer des versions spécialisées et optimisées des fonctions demandant un temps de calcul élevé. Ces fonctions optimisées et spécifiques à l'architecture matérielle seront disponible à l'utilisation dans l'ensemble de la plateforme logicielle. Tous les nouveaux algorithmes développés dans le cadre de ce projet devront pouvoir évoluer et exhiber une grande adaptabilité face au futurs noeuds de calcul du CSCS. Adaptabilité se basant sur la taille de la mémoire disponible, le nombre de noeuds de calcul et leur spécificités propres
- Continuer le développement du paradigme de programmation en flux de données haut niveau et son écosystème d'outils associé. Basé sur le standard existant, Dataflow Programming Standard ISO/IEX 23001-4 et utilisé jusqu'à présent principalement à la compression vidéo et streaming. Ce paradigme permettra de bénéficier des mêmes avantages qu'il offre dans le milieu vidéo mais cette fois-ci dans le cadre de la compression et du traitement de données génomiques. Ses avantages sont notamment ses implémentations portables, efficaces et hautement parallélisables.
De manière générale, les défis ci-dessus, font partie du domaine "data-intensive memory-bound applications" ou applications pour lesquelles la quantité de données, la mémoire nécessaire et les transferts d'informations sont les principaux problèmes rencontrés. De ce fait ils poussent de plus en plus la nécessité pour un matériel spécialisé (non-standard) et le développement d'algorithmes et de librairies haute performances.
Dans ce contexte l'institut REDS est spécialement impliqué dans l'acceleration pour la compression et décompression des données génomiques ainsi que la comparaison de séquences ARN/ADN. L'exploitation de matériel hautement configurable tels les FPGAs permet d'optimiser ces tâches.
Actuellement, afin de lire et d'analyser un génome humain, les machines de séquençage doivent diviser les chaines d'ADN en de nombreux fragments appelés séquences. Ces séquences sont ensuite partiellement lues par les machines dont les résultats sont enregistrés dans un fichier numérique. La machine effectue un nombre extrêmement nombreux de lectures de séquences (fragments d'ADN) afin de pouvoir garantir une bonne couverture de l'information contenue dans l’ADN d'un individu. De nombreuses séquences similaires ou identiques auront finalement été lues. Il est possible d'exploiter ce haut niveau de redondance contenu dans l’ensemble des lectures afin d'optimiser la compression.
Une étape fondamentale dans la compression de données génomiques est la détection et le groupement de séquences identiques (ou fortement similaires). Ce procédé, appelé clustering en anglais, permet ensuite de caractériser chaque groupe par leur points communs (partagés par tous les membres du groupe) et d'encoder seulement les différences que présentent les membres du groupe par rapport à leur points communs. Ce procédé de groupement peut prendre un temps extrêmement long dès qu'il doit être appliqué à un nombre important de séquences. Pour chaque génome lu il faudra trier et grouper plusieures dizaines de millions de séquences chacune composées de quelques centaines de bases. Réduire le temps d'exécution de ce procédé est un des défi principal dans le cadre du groupement de données génomiques.
Une base peut être représentée par une lettre : A,C,G et T cependant une cinquième lettre est utilisée: N, lorsque la machine n'arrive à déterminer de quelle base il s'agit. Il faut donc au minimum trois bits afin d'encoder le type d'une base. On peut remarquer que cette représentation sur 3bits n'est pas forcément optimale pour les processeurs actuels (qui travaillent avec des mots d'au minimum 8bits) cependant la technologie FPGA permet de travailler avec des mots de taille arbitraire et rend donc l'implémentation de fonctions traitant des bases sur 3bits très optimisées et possiblement massivement parallèles.

Le but de notre travail actuel est de développer un système capable d'accélérer drastiquement l'algorithme de groupement (clustering). Afin d'atteindre ce but le système sera mis en pratique sur un carte disposant de plusieurs FPGAs et mémoires à très hautes performances le tout relié à un PC via un lien PCI-Express.
Les caractéristiques principales du projet d'accélération du groupement de séquences ADN sont :
- La possibilité de traiter des séquences de longueur variable.
- L'utilisation de la mémoire disponible dans et autour de la FPGA afin de servir comme cache pour un accès extrêmement rapide aux séquences.
- L'incorporation d'éléments mémoires ultra modernes appelés HMC (Hybrid Memory Cube).
Afin de quantifier le gain en performances du développement de ce matériel hautement spécialisé il sera comparé à une implémentation software standard.